多么丰富多彩的世界
第9章 多么丰富多彩的世界
多么丰富多彩的世界强调设计-构建-测试周期的“构建”阶段。您将在此 BioBuilder 实验室活动中构建的合成生命系统是用紫色颜料或绿色颜料着色的细胞,具体取决于您选择运行的遗传程序。然而,颜色的强度很难提前预测。细胞会是苍白的还是军绿色的?深紫色还是浅紫色?让事情变得更加复杂的是,我们认识到每个基因回路可能会产生不同数量的颜色,具体取决于运行它的细菌菌株。组合组件产生的这种意想不到的、古怪的结果的正式术语是紧急行为。
如果你倾向于像科学家一样思考,这种突发行为可能会让你问:“如何解释我观察到的结果?”如果您倾向于像工程师一样思考,这些行为可能会让您觉得“呃”,然后开始定义优化系统性能的最佳方法,希望避免此类意外行为。这两种方法都有优点,并且科学和工程方法共同应该在未来带来更可靠的设计,并最大限度地减少我们在构建新的生命系统时看到的令人惊讶的行为数量。
在这个实验室中,生成紫色或绿色色素的 DNA 程序已经为您编写和组装,但您将通过将编码程序的 DNA 插入到一些不同的细菌底盘中来完成最终的构建步骤。 DNA 程序来自 2009 年国际基因工程机器 (iGEM) 项目,名为“E. chromi”,剑桥大学的学生设计并改造了大肠杆菌来生产一系列颜料。 您将构建多个颜色生成系统,以探索底盘如何影响设计的遗传程序的输出。由于颜料是肉眼可见的,因此您可以轻松确定机箱之间的颜色输出是否不同。
不过,在详细介绍实验之前,我们将对底盘设计和决策进行一般性讨论,并且我们将逐步介绍 E. chromi 项目,为您的调查提供一些背景信息。
机箱简介
正如汽车制造商在设计发动机时必须考虑整个汽车一样,合成生物学家也必须考虑他们正在构建的整个系统,包括他们设计的基因程序将在其中执行的细胞环境。就像路上有各种各样的汽车一样,细胞的大小、形状、细胞器和基本代谢功能也有很大差异。因此,为任何工程遗传程序选择最好的宿主细胞或底盘是设计过程中的重要一步。许多参数都会影响底盘的选择,包括特定于某些应用的一些考虑因素。然而,普遍关注的是实用性和安全性,我们将在以下几节中对此进行探讨。

实用程序以及如何设计它
在为合成生物学应用选择底盘时,研究人员会考虑通用实用性和利基实用性。一般效用是指具有普遍帮助性的品质;例如,在实验室环境中的快速增长。此外,普遍有用的底盘应该相对容易使用,这解释了为什么科学家依赖的许多常见模型生物(大肠杆菌、酿酒酵母等)对合成生物学家来说都是有吸引力的底盘。这些细胞拥有强大的工具,可以以合理且相当可靠的方式设计和操纵它们。
另一方面,利基效用是指使单元适合特定应用的品质。利基效用的一个例子是 BioBuilder 的 Eau That Smell 基因程序的细菌宿主。如果您熟悉这个实验装置,您会记得该项目的合成生物学家希望让细菌散发出香蕉或冬青的宜人香气。为了实现这一目标,他们通过从细胞色氨酸生物合成途径中删除一个基因,消除了所使用的大肠杆菌宿主产生的自然臭味,从而产生了一个“无臭”底盘来运行其产生气味的基因程序。
有两种互补的方法可以为每个可以想象的目的找到合适的底盘。在第一种方法中,我们可以称之为“最近的野生型生物”方法(见图 9-1),底盘是根据其执行类似于合成生物学家想要完成的任务的能力从自然界中识别出来的。一个新的系统。野生型生物体具有其自然界中物种最典型的特征,在这种方法中,野生型细胞的自然功能得到改进,使现有细胞越来越接近新系统中所需的行为。为了说明这种最接近野生型生物的方法,请考虑劳伦斯伯克利国家实验室辛格研究小组的工作。该小组的目标是改造富养罗尔斯通氏菌(Ralstonia eutropica),以生产丁醇和烯烃等生物燃料,有朝一日可能会取代石油基化学燃料。富养罗氏菌自然地将二氧化碳转化为储能聚合物,因此这些工程师认为这是一个有吸引力的底盘,并正在实验性地修改其自然途径,以便细胞产生生物燃料,而不是它们通常制造的聚合物。
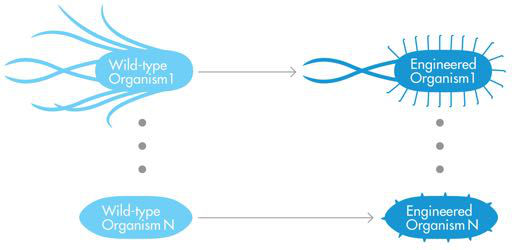
图 9-1 底盘选择的“最近野生型生物体”方法。野生型生物体1可以被改造为相对相似的工程化生物体1,其具有与亲本生物体相同的整体形状和多个鞭毛。类似地,野生型生物体 N 的形状与野生型生物体 1 略有不同,并且没有鞭毛,可以修改为工程生物体 N。对于新的野生型生物体中的每个实质性差异,都需要重复此过程。工程生物体所需的特性。
底盘设计的第二种方法从我们所说的“标准底盘”开始。在这种方法中,如图 9-2 所示,合成生物学家可能会选择更通用的底盘,该底盘要么具有最少数量的天然成分,要么易于理解且高度可工程化。简单的底盘本质上为合成生物学家提供了一块空白画布,并且至少在理论上可以成为具有完全不同的所需输出的各种工程系统的理想底盘。 Ginkgo Bioworks 的研究人员采用标准底盘方法,使用工程大肠杆菌来制造生物燃料。与我们之前考虑的例子不同,在该例子中,生物燃料生产是从天然聚合物工厂 R. eutropica 开始的,而大肠杆菌没有现有的人才可以推荐它们进行生物燃料生产。然而,大肠杆菌是该领域最接近标准细菌底盘的生物体。因此,Ginkgo Bioworks 的研究人员认为大肠杆菌是一个具有多种用途的有吸引力的平台,包括将二氧化碳和电能转化为异辛烷,异辛烷是一种非常适合美国现有运输燃料基础设施的液体燃料。
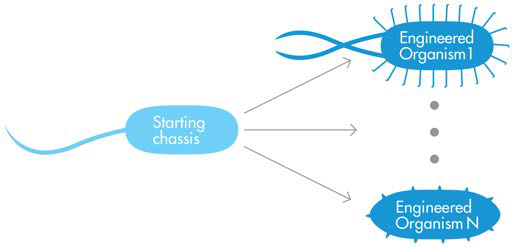
图 9-2 “标准机箱”方法。标准起始底盘简单且易于设计,因此合成生物学家可以将其用作工程有机体 1、工程有机体 N 以及介于两者之间的所有产品的先驱。
最近的野生型生物和标准底盘方法都有优点和缺点。例如,如果仅需要对现有生物体进行微小的改变即可获得所需的结果,则使用最接近的野生型生物体方法可能相对容易。然而,这种策略可能难以扩展,因为对于每个新的设计规范,设计人员都需要找到新的底盘。此外,使用现有生物体作为底盘可能会出乎意料地复杂,因为所有生物体都有自己的代谢途径和需求,其中任何一个都可能以令人惊讶的方式干扰所需的输出。
标准底盘方法避免了使用复杂生物体构建合成生命系统时出现的一些并发症。这种方法也比最接近的野生型生物方法更具可扩展性,因为它为设计者提供了一个通用的起点。借助通用的标准化机箱,设计人员可以将一种设计的工具重复用于另一种设计,至少在某种程度上是这样。天上掉馅饼的希望是“空白画布”细胞可以通过编程来生产药物、食品、组织或生物燃料,但目前这种希望相当牵强。目前,细胞还不能被抽象成一个不可知的画布来运行 DNA 程序。
然而,合成生物学家对计算机编程的成功感到鼓舞,因为这种跨平台工具确实存在。例如,Java等软件平台定义了“虚拟机”,使程序可以在任何操作系统上运行。 Java 虚拟机采用 Java 字节码,以便无论操作系统如何,它都以相同的方式工作。在这个类比中,Java 字节码类似于 DNA 序列,我们可以将 Java 虚拟机(加上操作系统)视为将该代码转换为操作的细胞机器。换句话说,我们可以将合成生物学的标准底盘视为生物学的Java虚拟机。
然而,想象每个合成生物学应用都有一个标准底盘还不现实,因此在最近的野生型和标准底盘方法之间达成了妥协。在这种妥协中,一个小的底盘菜单将作为合成生物学家的起点,他们可以为任何给定的应用领域从几种不同的细胞类型中进行选择。在这种情况下,可能存在一两个用于细菌应用的可靠标准底盘,以及另外一两个用于在哺乳动物细胞中运行的遗传程序的标准底盘。尽管该技术仍在开发中,但这种细胞底盘工程的混合方法也许更有可能为合成生物学家在短期内取得成功提供可用的工具。
安全性以及如何设计它
在选择用于运行工程 DNA 程序的机箱时,您必须始终考虑安全性。具体来说,研究人员必须考虑他们在使用合成生命系统时的个人安全、实验室环境的安全,以及如果他们的工程生物体进入更广阔的世界可能发生的任何伤害。
现有的指南和法规在很大程度上保护了研究人员及其工作所在实验室社区的人身安全。随着重组 DNA 技术的出现,这些法规于 20 世纪 70 年代开始实施。它们通常被认为是成功的,但当时引起了激烈的争论,这些争论为今天的合成生物学提供了许多相关的教训。有关指南及其发展的更详细说明,请参阅生物伦理学基础章节。
除了从基因工程领域继承的监管工作之外,合成生物学家还必须仔细考虑将合成生命系统安全地引入我们复杂的环境中。理想情况下,应该有一种通用方法来最大限度地减少和遏制可能逃逸或故意部署到实验室外环境中的生物体造成的任何潜在损害。由于它们携带额外的遗传负担,大多数合成生物体相对于其天然竞争者来说适应性下降,这已经降低了工程生物体在野外持续存在或造成重大损害的可能性。然而,依赖这种不平等似乎是不明智的,并且在合成生物学家规划他们的系统时正在设计额外的安全措施。
相反,合成生物学家正在尝试进行安全设计。 “设计安全”的一个根本例子是合成生物学研究,以设计与天然存在的 DNA 不同的解码方式的正交 DNA。由于DNA可以在生物体之间转移,而且这种转移相对常见,因此人们担心如果工程生物体从自然存在的生物体中获取DNA,反之亦然,可能会产生潜在的后果。然而,如果工程生物体表达的正交 DNA 无法在原始宿主之外正确解码,那么这些可能的危险就会最小化。任何使用改变的遗传密码的底盘都无法实现从外部环境获得的新的且具有潜在危险的遗传功能,并且如果将来自工程生物体的基因转移到自然存在的环境中,则无法实现它们有机体。
合成生物学家仍处于此类研究的早期阶段,但 2011 年《科学》杂志上发表的一篇论文显示了一些有希望的结果。由哈佛大学 George Church 博士实验室领导的研究小组通过用同义 TAA 终止密码子替换他们在 460 万个碱基对基因组中发现的所有 TAG 终止密码子,对大肠杆菌进行了“重新编码”,如图 9 所示-3。将这组看似“无声”的变化结合起来是一项技术杰作,促使团队开发出新的、非常有用的基因组操作工具,称为“MAGE”和“CAGE”。然而,与本次讨论更相关的是,与天然菌株相比,所得底盘具有两个安全优势。首先,新菌株中的所有编码序列仍然可以转录和翻译,但因为它们都没有使用 TAG 密码子,所以可以重新调整该密码子,将替代氨基酸添加到新的 DNA 程序中。 然后可以通过使其依赖于研究人员提供的一些非天然氨基酸(而不是自然提供的)来设计新生物体以确保安全。其次,TAG密码子不再在重新编码的大肠杆菌中充当终止密码子。因为Church研究小组还删除了在任何TAG处终止翻译的释放因素。因此,如果重新设计的细菌拾取任何使用 TAG 终止密码子的外部 DNA,所得的多肽链将失去功能,因为核糖体将继续读取超过终止密码子的内容。 必须做进一步的工作才能使工程 DNA 真正成为现实。与自然发生的遗传物质正交,但“安全设计”的早期成功是一个值得考虑的很好的模型,随着努力的推进。
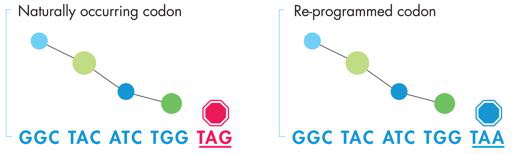
图 9-3 一种生成正交 DNA 的方法。左侧天然存在的 DNA 序列编码四个氨基酸,由序列上方的蓝色和绿色球表示,以 TAG 终止密码子结尾。通过将 DNA 天然存在的 TAG 终止密码子(左)转换为 TAA 终止密码子(右),蛋白质产物不会发生变化,但表达该遗传密码的细胞可以通过将 TAG 密码子重新分配给非天然氨基酸来重新设计成正交基因组。
E. chromi iGEM 项目的背景

剑桥大学团队在 2009 年的 iGEM 项目中设计、指定并构建了一套生物传感器,团队将其称为 E. chromi。鉴于生活世界中存在各种各样的自然颜色(橙色胡萝卜、紫色花朵等),该团队决定使用颜色作为其系统的输出。它还决定为特定金属化合物制造传感器,包括砷、汞和其他重金属,这些金属在一些国家一直是污染问题。 这些环境污染物输入将触发肉眼可见的基于颜料的输出,以便轻松、立即部署。该设计是对之前使用 pH、电导和荧光等输出的 iGEM 生物传感器项目的改进,因此需要额外的步骤来检测输出信号。这些颜料还提供了视觉多样性,激励团队构建一种活的“量油尺”,可以用一个样本拭子以彩色方式报告多种污染物,如图 9-4 所示。
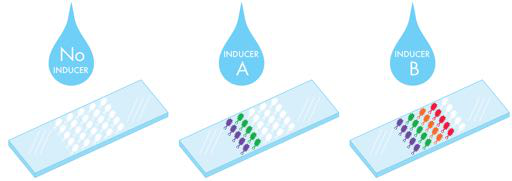
图 9-4 检测单个水样中多种污染物的量油尺。量油尺上会发现表达不同基因程序的细菌,这些基因程序能够感知污染物并产生颜色响应。当暴露在没有任何诱导剂的水中时(左),不会产生颜色。当暴露于含有两种污染物的混合物(中心)的水中时,会产生两种相应的颜色。如果水中含有四种污染物(右),就会产生四种颜色。
除了检测特定污染物的存在之外,研究小组认为估算每种污染物的浓度也很有用。 通过开发一系列控制系统灵敏度的“调谐器”设备,该团队可以生成每个颜色生成系统的变体,以便系统仅当污染物存在于特定浓度范围内时才会改变颜色。砷浓度低的水样只会触发低灵敏度调谐器,并将信号传递到该变体中的颜色生成装置。含有高浓度砷的水样将触发低、中、高灵敏度调谐器,并触发所有三种变体的颜色。通过最终的系统(图 9-5),该团队可以将其收集的变体暴露于水样中,并根据哪些变体产生颜色、哪些不产生颜色,确定存在哪些污染物以及浓度。
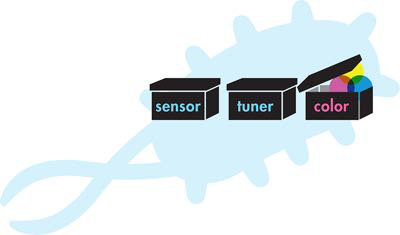
图 9-5 活体生物传感器的系统级设计。用于检测环境污染物并报告其浓度的电池的黑盒设计理念。
关于他们的设备

为了找到编码鲜艳颜色的 DNA 序列,iGEM 团队挖掘了文献,最终确定了三种能够产生独特色素的自然存在的细菌系统。这些系统如表 9-1 所示。
表 9-1 产生生动色彩的天然来源

由于该团队想要多种颜色,因此他们将所有这些来源作为潜在的设备,并发现其中一些更适合某些应用。例如,黑色素由单个基因编码,这使得通过已建立的基因调控方法相对容易控制输出,但该系统还需要在培养基中添加铜和酪氨酸,从而使菌株的生长条件变得复杂表达这种颜色。另外两个色素家族是通过多个基因产物的作用产生的,这使得它们的调节更加复杂,但产生这些色素的细胞更容易生长,因为颜色是在生长培养基中没有任何不寻常的化合物的情况下产生的。
为了部署系统的设备,iGEM 团队还必须考虑用于生产紫罗兰素和类胡萝卜素色素的不同机制。类胡萝卜素装置首先使用一种途径产生红色素,然后使用下游酶将红色素的一部分转化为橙色素。相比之下,紫罗兰素装置依靠分支代谢途径来产生绿色和紫色,如图 9-6 所示。
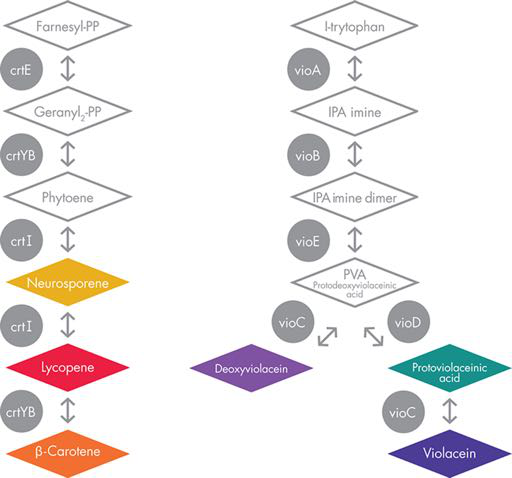
图 9-6 色素生成代谢途径。左侧显示类胡萝卜素途径,右侧显示紫罗兰素途径。菱形代表不同的细胞中间体,圆圈代表负责化学反应的酶。有色化合物以其近似颜色表示。
零件和设备
BioBuilder 多彩世界活动使用紫罗兰素系统的成分,根据相对较小的基因变化,可以产生绿色或紫色色素。通常,该色素生成系统的输入是氨基酸色氨酸,通过五种酶(VioA、VioB、VioC、VioD 和 VioE,但不是按此顺序!)的作用,产生一种称为紫罗兰素的紫色色素。然而,当 vioC 基因被去除时,该途径分支被阻断,最终无法转化为紫草素,因此系统的终点反而变成了原紫草酸,一种深绿色的色素。 您可以通过检查代谢途径来推断去除 vioC 基因的效果(图 9-6)。这种行为也可以使用逻辑门和真值表的工程形式来说明,如图 9-7 所示。
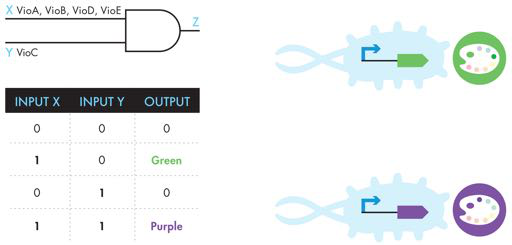
图 9-7 紫罗兰素相关系统的不同表示。逻辑门表示(左上)显示输入 X(VioA、B、D 和 E)和输入 Y (VioC) 必须如何存在才能生成紫色输出 Z。修改后的真值表(左下)显示输入 X单独可以创建绿色输出,而单独输入 Y 不会生成输出,输入 X 和输入 Y 生成紫色输出。右侧的遗传示意图显示,绿色生成装置(由 VioA、B、D 和 E 组成)上游的启动子产生绿色色素,紫色生成装置(由 VioA、B、D 和 E 组成)上游的启动子产生绿色色素。 B、C、D 和 E,产生紫色颜料。
额外阅读和资源
- Changhao, B. 等人开发用于富养罗尔斯通氏菌的广泛宿主合成生物学工具箱及其在工程碳氢化合物生物燃料生产中的应用。微生物细胞工厂 2013;12:107。
- Ginkgo Bioworks Foundry (http://ginkgobioworks.com/foundry/ )。
- 拉乔伊,MJ。等人。基因组重新编码的生物体扩展了生物功能。科学2013;342(6156):357-60。
- E. chromi 网站 (http://www.echromi.com/ )。
多么丰富多彩的世界实验室
在本实验室中,将使用细菌转化技术将一些质粒 DNA 引入两种细胞类型。通过比较两种不同大肠杆菌菌株中相同基因程序的功能,探索了底盘的工程概念。无菌技术和固态微生物培养是本实验室强调的生物技术技能。
设计选择
在我们对 E. chromi 项目的讨论中,我们看到 iGEM 团队对系统的性能做出了一些假设。特别是,团队成员推测,当暴露于给定浓度的金属污染物时,颜色生成设备将可预测地生成可见的调色板。然而,研究小组意识到,每种色素生成装置都有其特殊性。例如,黑色素需要补充介质。为了解决这些特性,它对多种细菌菌株进行了实验,旨在找到运行每台设备的“最佳”菌株。
在本实验中,您将了解 iGEM 团队项目的这方面内容。 您将比较两个不同机箱中的两个颜色生成设备,以研究是否可以获得可靠的颜色输出。
实验问题
您将在两种常见的实验室大肠杆菌菌株(K12 菌株和 B 菌株)中测试紫色和绿色颜色生成装置。这两种菌株通常用于研究细菌细胞的行为和执行分子生物学技术。它们在实验室环境中存在了很长时间,以至于它们的许多基因发生了突变,这些突变不再是它们在狭窄的条件下生存所必需的。结果,它们几乎失去了在实验室环境之外茁壮成长的能力。鉴于对这些细胞的描述,它们似乎为细菌合成生物学的标准底盘提供了开端,至少在安全方面是这样。在本实验室中,您将通过测试它们是否产生可靠、可重复的行为来调查它们是否也接近实现标准机箱的“实用”要求。
要进行此实验,您需要将携带紫色和绿色颜色生成装置的基因程序转换到每种细菌菌株中(图 9-8)。这个过程涉及的步骤包括修补菌株以培养您需要的细胞,然后用盐溶液处理细胞,以便它们能够吸收 DNA。您将感受态大肠杆菌菌株与编码颜色生成装置的 DNA 混合,然后将细胞暴露在高温下一小段时间。这种热休克有助于细胞从环境中吸收 DNA。在最后的实验步骤中,您将在转化混合物中添加一些新鲜培养基以帮助细胞恢复,然后将转化的细胞铺板到装有琼脂固化培养基的培养皿上,其中还含有抗生素氨苄青霉素。编码颜色生成装置的 DNA 也编码氨苄青霉素抗性基因,因此唯一可以在平板上生长的细胞是那些具有颜色生成装置的细胞。
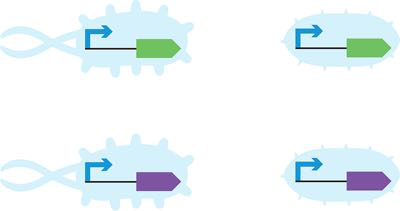
图 9-8 不同机箱中的颜色生成设备。您将把相同的设备插入不同的机箱(左与右)并研究它们的行为。
作为阴性对照,您将在转化过程中加入不含质粒的样品。该阴性对照预计不会在含有氨苄青霉素的平板上生长,因为它不包含氨苄青霉素抗性基因。如果观察到生长,您就知道您的程序或材料存在某些问题,无法仅选择已成功用质粒转化的细胞。一个潜在的原因可能是平板中的氨苄青霉素随着时间的推移而降解。包含对照对于良好的实验设计至关重要,因为它使您可以隔离变量,并可以为以后尝试解决实验中遇到的问题提供信息。
您收集的数据将基于对板的目视检查——您的眼睛是您需要的唯一精美的检测设备。经过一晚的生长,每个在转化和抗生素暴露中幸存下来的细胞都有时间生长成肉眼可见的细胞集落。您将对菌落进行计数以确定转化效率,并且您将记录不同板上菌落的颜色、形状和大小,以确定选择作为底盘的菌株是否会影响系统的输出。
入门
表 9-2 详细介绍了“多彩世界”实验中比较的两种质粒。
表 9-2 多么丰富多彩的世界质粒和菌株描述
| 质粒 | 注册质粒# | 质粒描述 |
|---|---|---|
| pPRL | BBa_K274002 | pUC18 质粒骨架,AmpR |
| pGRN | BBa_K274004 | pSB1A2 质粒骨架,AmpR |
4-1: NEB 大肠杆菌 K12 ER2738:F’proA+B+ lacIq Δ(lacZ)M15 zzf::Tn10(TetR)/ fhuA2 glnV Δ(lac-proAB) thi-1 Δ(hsdS-mcrB )5
4-2: NEB 大肠杆菌 BL21 C2523:fhuA2 [lon] ompT gal sulA11 R(mcr-73::miniTn10– TetS)2 [dcm] R(zgb-210::Tn10–TetS) endA1 Δ(mcrC-mrr)114::IS10
该实验室需要两种大肠杆菌菌株:4-1(K-12 型)和 4-2(B 型)。为了获得足够数量的细菌来进行转化,您需要将这些菌落重新接种为补片。每个贴片将为一个实验室小组提供足够的细菌,一个平板上最多可容纳六个贴片。
- 使用无菌接种环或牙签,将细菌菌落从其中一个培养皿转移到新的 Luria Broth (LB) 琼脂(非 LB+amp)培养皿中,为每种菌株绘制 1 cm x 1 cm 的正方形。以这种方式绘制的每个正方形都会产生足够的细胞来用两个质粒进行转化。
- 对转化实验室所需的每种菌株重复上述步骤。
- 将培养皿置于 37°C 培养箱中过夜。
实验室协议
- 将两个小微量离心管标记为“4-1”和“4-2”。
- 将 200 μl CaCl_2 转化溶液移入每个管中,然后将管置于冰上。
- 使用无菌木钉或接种环刮起一整块标有“4-1”的细胞(不包括它们生长的琼脂!),然后将细胞旋入装有冷 CaCl_2 的管中少量琼脂可能会被转移,而不会影响您的实验。如果您有涡旋,您可以通过涡旋一分钟来使细胞重新悬浮。如果没有涡流,轻轻弹动并翻转管子。
- 重复,使用不同的无菌木钉刮掉标记为“4-2”的细胞块。如果可能的话,短暂涡旋。一些细胞团保留在该溶液中是可以的。
- 在准备用于转化的 DNA 时,将这些感受态细胞置于冰上。
- 在每个质粒的微量离心管中取出两等份,总共四个样品(2x 紫色生成装置质粒,pPRL,2x 绿色生成装置质粒,pGRN)。每个等份含有 5 ul DNA。 DNA 的浓度为 0.04 μg/μl。当您在实验结束时计算转化效率时,您将需要这些值。
- 将其中一支 pPRL 管标记为“4-1”。将另一个 pPRL 管标记为“4-2”。确保标签可读。将管子放入冰桶中。
- 将其中一支 pGRN 管标记为“4-1”。将另一个 pGRN 管标记为“4-2”。确保标签可读。将管子放入冰桶中。
- 用感受态 4-1 菌株轻弹试管,然后将 100 μl 细菌移入标有“pPRL, 4-1”的试管中,再将 100 µl 细菌移入标有“pGRN, 4-1”的试管中。轻弹以混合管子并将其放回冰中。将剩余的少量 4-1 菌株保存在冰上。
- 用感受态 4-2 菌株轻弹试管,然后将 100 μl 移液至标有“pPRL, 4-2”的试管中,再将 100 μl 移入标有“pGRN, 4-2”的试管中。轻弹混合并将它们以及剩余体积的感受态细胞储存在冰上。
- 将 DNA 和细胞置于冰上 5 分钟。
- 当您的 DNA 和细胞孵化时,您可以标记您需要的六个培养皿的底部(介质侧)。标签应标明您使用的菌株(“4-1”或“4-2”)以及转化它们所用的 DNA(“pPRL”、“pGRN”或“无 DNA 对照”)。
- 将试管置于 42° 温度下精确热激 90 秒(使用计时器)。
- 90 秒结束时,将管子移至室温下的架子上。
- 将 0.5 ml 室温 LB 添加至试管中。盖上盖子,翻转试管以混合内容物。
- 使用无菌涂布器或无菌珠子,将 200–250 μl 转化混合物涂布到 LB+氨苄青霉素琼脂培养皿的表面。
- 盖上盘子并放置一分钟。然后,将盘子翻过来。培养皿将倒置存放,以防止冷凝水滴到细菌上。
- 将培养皿琼脂面朝上,在 37°C 下孵育过夜。